Từ năm 2014 đến năm 2019, nhóm nghiên cứu của TS. Nguyễn Thị Hồng Thương tại Trường Đại học Khoa học Tự nhiên đã thực hiện đề tài: “Phân lập và khảo sát các enzyme methylketone synthase 2 (MKS2) mới từ một số loài thực vật thuộc họ Brassicaceae, Solanaceae và Fabaceae”.
Đề tài hướng đến thực hiện mục tiêu: Phân lập được gene mã hóa methylketone synthase 2 (MKS2) hay còn gọi là βketoacyl-ACP thioesterase ở một số loài thực vật thuộc họ Brassicaceae, Solanaceae, và Fabaceae; xác định được hoạt tính thioesterase của các protein được mã hóa bởi những gene này nhằm bổ sung vào bộ sưu tập MKS2 những enzyme mới, mỗi enzyme có khả năng thủy phân một (hoặc một vài) cơ chất 3-ketoacyl-ACP khác nhau về chiều dài và mức độ bão hòa của khung carbon; từ đó, hướng đến tổng hợp được đa dạng các hợp chất methylketone đáp ứng tiềm năng ứng dụng rộng rãi của chúng trong bảo vệ thực vật, trong công nghiệp thực phẩm, mỹ phẩm và trong lĩnh vực sản xuất năng lượng sinh học.
Sau năm năm nghiên cứu, đề tài đã đạt được một số kết quả sau:
- Bằng cách sử dụng công cụ tìm kiếm TBLASTN và trình tự truy vấn ShMKS2 để tra cứu cơ sở dữ liệu bộ gene hoặc EST của một số loài thực vật được chọn làm đối tượng nghiên cứu nhằm tìm kiếm các ORF hoặc các phân đoạn cDNA mã hóa cho protein tương đồng với trình tự ShMKS2 đã biết, nhóm nghiên cứu đã xác định được các trình tự MKS2 tương đồng với ShMKS2 ở một số loài thực vật khác nhau thuộc họ Solanaceae (Cà chua Solanum pimpinellifolium, Ớt Capsicum annuum, Cà tím Solanum melongena, Thuốc lá Nicotiana tabacum, Khoai tây Solanum tuberosum), họ Fabaceae (đậu nành Glycine max, đậu cô-ve Phaseolus vulgaris), họ Brassicaceae (Arabidopsis thaliana).
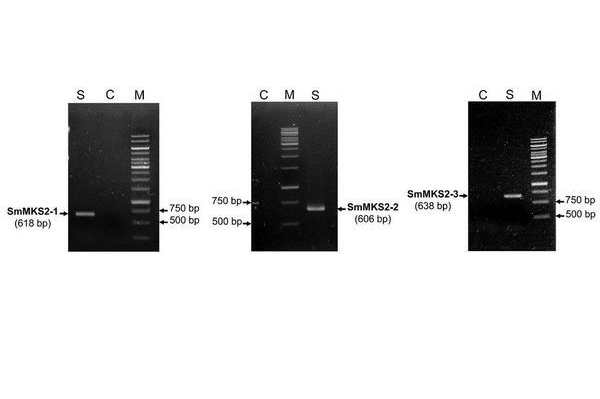
- Dựa trên trình tự được dự đoán bằng công cụ tin-sinh học, các nhà khoa học đã thiết kế mồi phân lập được các cDNA hoàn chỉnh mã hóa cho các MKS2 bao gồm: GmMKS2-X1, GmMKS2-X2 và GmMKS2-X3 từ đậu nành Glycine max (họ Fabaceae); SmMKS2-1, SmMKS2-2 và SmMKS2-3 từ cà tím Solanum melongena (họ Solanaceae); CaMKS2 từ ớt Capsium annuum (họ Solanaceae); NtMKS2-1 và NtMKS2-2 từ thuốc lá Nicotiana tabacum (họ Solanaceae); StMKS2-1và StMKS2-2 từ khoai tây Solanum tuberosum (họ Solanaceae).
- Dòng hóa được các trình tự cDNA MKS2 trên vào vector pGEM-T, giải mã trình tự và so sánh với trình tự tương ứng được dự đoán bằng công cụ tin-sinh học.
- Tạo dòng được các gene MKS2 phân lập ở trên vào vector biểu hiện pETDuet-1 và biến nạp vector mang gene tái tổ hợp này vào tế bào vi khuẩn E. coli C41 (DE3) để khảo sát hoạt tính enzyme in vivo của các protein MKS2 trong E. coli.
- Phân tích in silico trình tự promoter của các gene GmMKS2, SmMKS2-1, SmMKS2-2 và SmMKS2-3, dự đoán sự hiện diện của các yếu tố điều hòa cis trên trình tự promoter của các gene này.
- Khảo sát sự biểu hiện phiên mã của hệ gene MKS2 trong các mô khác nhau và trong đáp ứng với các tín hiệu stress ở hai loài đậu nành G. max và cà tím S. melongena.
Các kết quả nghiên cứu mới và có giá trị khoa học cao đã được nhóm nghiên cứu trình bày trong 3 bài báo đăng trên Tạp chí Khoa học uy tín trong nước, 3 báo cáo Kỷ yếu Hội thảo trong nước/quốc tế, và bài báo có tiêu đề “Characterization of Solanum melongena Thioesterases Related to Tomato Methylketone Synthase 2” công bố trong Tạp chí Genes thuộc hệ thống ISI (SCIE, hạng Q1 theo Scimago).
Có thể tìm đọc báo cáo kết quả nghiên cứu (mã số 16798/2019) tại Cục Thông tin Khoa học và Công nghệ Quốc gia.
N.P.D (NASATI)