Chu Đức Hà1, Phùng Thị Thu Hương1, Phạm Bích Ngọc2, Lê Thị Ngọc Quỳnh3,
Lê Hùng Lĩnh1, Phạm Xuân Hội1, Lê Tiến Dũng4
1Viện Di truyền Nông nghiệp, VAAS
2Viện Công nghệ Sinh học, VAST
3Khoa Hóa và Môi trường, Trường Đại học Thủy lợi
4Khoa Công nghệ Sinh học, Trường Đại học Nguyễn Tất Thành
Chỉnh sửa hệ gen với hệ thống CRISPR/Cas đã trở thành một trong những công cụ đắc lực nhằm cải thiện các tính trạng của cây trồng. Về bản chất, hệ thống CRISPR/Cas cho phép can thiệp vào gen tại những vị trí có định hướng. Cho đến nay, khoảng 24 loài cây trồng, với ít nhất 193 gen đã được báo cáo chỉnh sửa thành công nhằm cải thiện những đặc tính liên quan đến quá trình trao đổi chất, khả năng chống chịu bất lợi và các yếu tố cấu thành năng suất. Ở lúa gạo (Oryza sativa), nỗ lực của các nhà khoa học cũng đã được ghi nhận trong việc cải biên các gen kháng thuốc diệt cỏ, asen (ALS, ARM1) hoặc quy định năng suất (AAP3, GS3, DEP1, GW2, PYL1, PYL4, PYL6, Gn1a). Bài viết tổng hợp những thành tựu của chỉnh sửa hệ gen trên lúa gạo, từ đó đưa ra thảo luận một số ý kiến nhằm xây dựng một chiến lược nghiên cứu dài hạn cho chỉnh sửa hệ gen trên cây trồng nói chung và lúa gạo nói riêng.
Mở đầu
Những tiến bộ nhanh chóng của kỹ thuật chỉnh sửa hệ gen (genome editing – GE) đã tạo ra một cuộc cách mạng thực sự trong nghiên cứu chức năng gen và cải thiện di truyền ở thực vật. Trong những thập niên trước, bài toán can thiệp vào tính di truyền ở thực vật từng được thực hiện một cách rộng rãi thông qua các phương pháp lý hóa như sử dụng tia gamma hoặc hóa chất (ethyl methanesulfonate) và phương pháp sinh học (T-DNA, transpose) để tạo ra các đột biến ngẫu nhiên trong hệ gen. Giờ đây, với sự ra đời của kỹ thuật GE, phổ biến hơn cả là hệ thống CRISPR/Cas (Clustered regularly interspaced short palindromic repeats, Cas) đã cho phép cải biến phân tử ADN mục tiêu một cách chính xác và có chủ đích.
Đến nay, cây trồng được chỉnh sửa bằng CRISPR/Cas9 được cho là vượt qua nhiều rào cản về xếp loại cây trồng biến đổi gen [1]. Gần một thập kỷ qua, hàng ngàn ấn phẩm về chủ đề GE ở thực vật đã được đăng tải, số bài báo mới và các ứng dụng mới được công bố đang tăng lên từng ngày.
Về mặt lý thuyết, GE lợi dụng hệ thống sửa chữa ADN của tế bào để tạo ra những thay đổi nhỏ trong trình tự ADN đích, thông qua việc sử dụng enzyme nuclease tổng hợp để tạo ra các đứt gãy ADN sợi đôi (DSB) tại vị trí được định hướng trong hệ gen. Các đứt gãy này sẽ được tế bào sửa chữa thông qua cơ chế sửa chữa ghép nối không tương đồng (non-homologous end-joining – NHEJ) hoặc chèn thêm một trình tự ADN vào vị trí đứt gãy theo cơ chế sửa chữa tái tổ hợp tương đồng (homologous recombination – HR). NHEJ là cơ chế sửa chữa DSB phổ biến nhất trong các tế bào thực vật, cơ chế này hoạt động hiệu quả nhưng dễ xảy ra lỗi (sửa chữa không chính xác), trong khi đó cơ chế HR kém hiệu quả hơn nhưng có độ chính xác cao. Kết quả của quá trình sửa chữa DSB có thể là sửa chữa chính xác hoặc xuất hiện các đột biến indel (thay thế, mất hoặc chèn gen), tùy vào con đường được sử dụng là HR hay NHEJ. ZFNs (Mega-nuclease, zinc finger nucleases), TALENs (transcription activator-like effector nucleases) là những công cụ đầu tiên của kỹ thuật GE, được sử dụng thành công trong chỉnh sửa hệ gen của nhiều đối tượng thực vật, bao gồm ngô, đậu tương và thuốc lá.
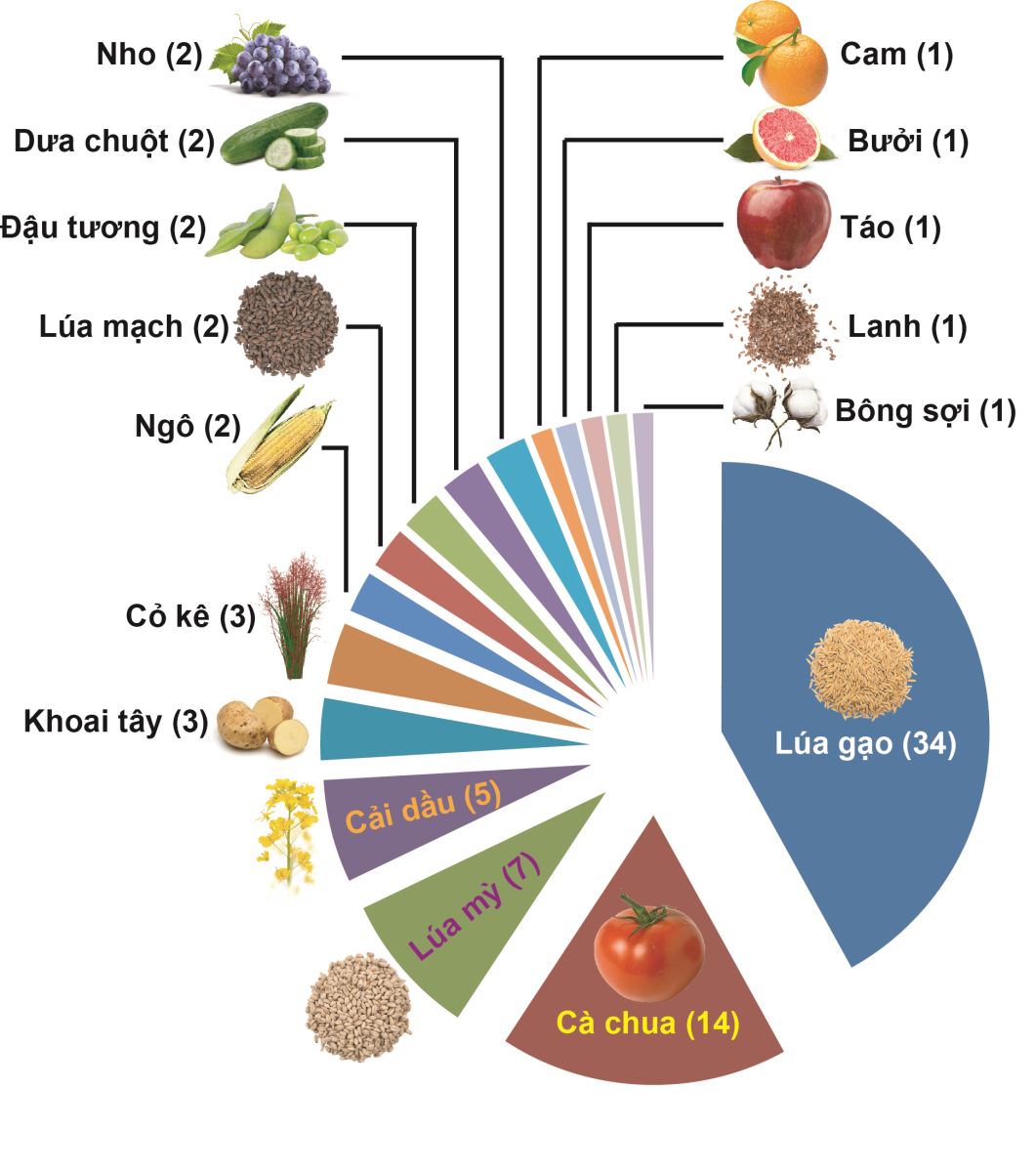
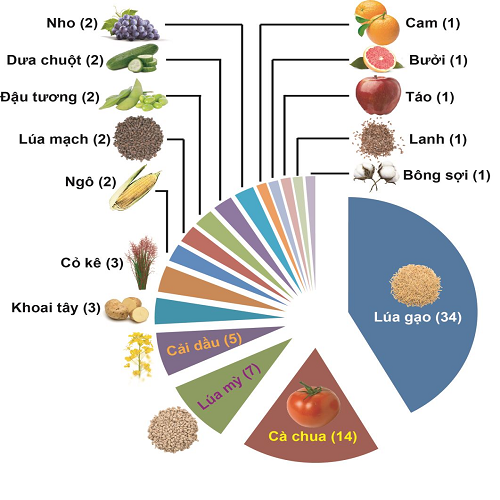
Hình 1. Số gen ở cây trồng được chỉnh sửa bằng hệ thống CRISPR/Cas9 với mục đích cải tiến di truyền (giai đoạn từ tháng 8/2013-8/2018) [2].
Công nghệ GE thực sự bùng nổ từ năm 2012 khi phức hợp CRISPR/Cas được ứng dụng để tạo DSB trên hệ gen. Hệ thống CRISPR/Cas trở thành cách tiếp cận mới thay thế các công cụ GE trước kia, cho phép chỉnh sửa hệ gen của sinh vật nhân chuẩn một cách chính xác, linh hoạt hơn và mang lại hiệu quả kinh tế hơn. Đến nay, 24 đối tượng cây trồng, với ít nhất 193 gen, đã được chỉnh sửa bằng hệ thống CRISPR/Cas [2]. Các nhà khoa học đã tập trung chủ yếu vào những cây lương thực, như lúa gạo, lúa mì, cỏ kê, ngô, lúa mạch; một số loại cây rau, như cà chua, khoai tây, dưa chuột; cây ăn quả như táo, nho, bưởi, cam; cây họ Đậu (đậu tương) và cây công nghiệp chính như bông, lanh. Những tiến bộ lớn nhất đã đạt được là trên lúa gạo (34 gen), cà chua (14 gen), lúa mì (7 gen), cải dầu (5 gen), các loại cây trồng còn lại chỉ có một gen được chỉnh sửa (cam, bưởi, táo, lanh, bông sợi) hoặc hai gen (ngô, lúa mạch, đậu tương, dưa chuột, nho) (hình 1). Trong đó, phần lớn cây trồng được chỉnh sửa gen với mục đích cải thiện khả năng chống chịu (thuốc diệt cỏ, kháng asen) hoặc năng suất, chất lượng.
Những thành công trong chỉnh sửa hệ gen lúa gạo
Lúa gạo được xem là cây mô hình một lá mầm quan trọng cho các nghiên cứu chức năng gen, đặc biệt là trong hướng đánh giá tính hiệu quả của các công cụ GE. Công trình tiếp cận với công cụ GE đầu tiên ở cây lúa gạo được báo cáo vào năm 2012 với việc sử dụng TALENs nhằm mục tiêu chỉnh sửa gen Os11N3 (OsSWEET14) – một gen “nhiễm” quan trọng mã hóa cho protein vận chuyển đường tham gia thúc đẩy quá trình xâm nhiễm của vi khuẩn gây bệnh bạc lá trên cây lúa để tạo dòng lúa kháng bệnh bạc lá [3]. Tháng 8/2013, lần đầu tiên hệ gen lúa gạo được chỉnh sửa bằng CRISPR/Cas [4], kể từ đó rất nhiều nghiên cứu tương tự đã được thực hiện [5]. Theo số liệu từ Scopus, trong gần 2 năm trở lại đây, lúa gạo là đối tượng được chỉnh sửa gen nhiều nhất, với tổng số 109 gen, trong đó chủ yếu là gen liên quan đến các đặc tính năng suất (AAP3, GS3, DEP1, GW2, PYL1, PYL4, PYL6 và Gn1a) và chống chịu (như ALS và ARM1) (bảng 1).
Bảng 1. Các gen được chỉnh sửa bằng hệ thống CRISPR/Cas trên lúa gạo.
| Gen | Chức năng | Dạng đột biến | Kết quả | Đối tượng | Phương pháp chuyển gen | Hiệu suất chỉnh sửa (%) | Nguồn |
| AAP3 | Vận chuyển amino acid | Bất hoạt | Tăng năng suất (tăng số chồi và số nhánh trên bông) |
Zhonghua 11 Kongyu 131
|
Agrobacterium
|
N/A | [6] |
| ALS | Enzyme sinh tổng hợp acid amin | Thay thế | Kháng thuốc diệt cỏ | Nipponbare |
Tế bào trần/ Agrobacterium
|
16,88 | [7] |
| ALS | Enzyme sinh tổng hợp acid amin | Thay thế | Kháng thuốc diệt cỏ | Nipponbare |
Agrobacterium
|
18,3 | [8] |
| ARM1 | Mã hóa R2R3 MYB | Bất hoạt | Tăng tính kháng Asen | Nipponbare
Dongjing |
Agrobacterium
|
N/A | [9] |
| BADH2 | Quy định mùi hương hạt | Bất hoạt | Mùi hương | Xidao |
Agrobacterium
|
70 | [10] |
| DEP1 | Quy định cấu trúc bông và chiều cao cây | Bất hoạt đa gen | Độ dựng và chắc của bông; bán lùn | Nipponbare
|
Agrobacterium
|
83 | [11] |
| EP3 | Quy định kiểu hình bông lúa | Độ dựng (thẳng) của bông | 50 | ||||
| GS3 | Quy định kích thước hạt | Tăng kích thước hạt | 100 | ||||
| GW2 | Quy định trọng lượng hạt | Tăng trọng lượng hạt | 67 | ||||
| Gn1a | Quy định số lượng hạt | Tăng số hạt trên cụm hoa | 97 | ||||
| BADH2 | Quy định mùi hương hạt | Mùi thơm | 81 | ||||
| Hd1 | Làm chậm thời gian ra hoa | Ra hoa sớm | 78 | ||||
|
SBEIIb
|
Enzyme phân giải tinh bột | Bất hoạt | Tăng amylose và tinh bột kháng | Kitaake | Agrobacterium |
40 |
[12] |
| FAD2-1 | Mã hóa acid béo desurase loại 2 | Bất hoạt | Tăng hàm lượng acid oleic; giảm hàm lượng acid linoleic |
Nipponbare
|
Agrobacterium
|
N/A | [13] |
| HAK1 | Vận chuyển ion K+ | Bất hoạt | Giảm hàm lượng ion Cs+ |
Nipponbare
|
Agrobacterium
|
83 | [14] |
| Hd2, Hd4,
Hd5 |
Kiểm soát thời gian ra hoa | Bất hoạt | Ra hoa sớm | Longdao16
Longdao18 Daohuaxiang2 |
Agrobacterium
|
77,8 | [15] |
| LCT1 | Vận chuyển Cd | Bất hoạt | Giảm vận chuyển Cd tới hạt | Xidao |
Agrobacterium
|
70 | [10] |
| Nramp5
|
Gen vận chuyển kim loại | Bất hoạt | Giảm vận chuyển Cd tới hạt | Huazhan
Longke 638S |
Agrobacterium
|
82,4 | [16] |
| NRT1.1B | Vận chuyển Nito | Thay thế | Tăng hiệu quả sử dụng đạm | Zhonghua 11 | Súng bắn gen | 6,72 | [17] |
Enzyme acetolactate synthase (ALS) xúc tác bước đầu tiên của quá trình sinh tổng hợp các amino acid chuỗi nhánh Leucine, Isoleucine và Valine. ALS là enzyme đích của ít nhất 4 nhóm thuốc diệt cỏ có cấu trúc riêng biệt (sulfonylureas, imidazolinones, triazolopyrimidine sulfonamides và pyrimidinyl carboxy). Butt và cộng sự đã gây đột biến và sửa chữa ALS bằng công cụ GE thông qua trung gian cgRNA để tạo ra cây lúa kháng thuốc diệt cỏ bispyribac natri (BS). Locus ALS ở Oryza sativa L. ssp. japonicacv. Nipponbare (LOC_Os02g30630) đã được chỉnh sửa bởi hệ thống cgRNA/Cas9 thông qua con đường HDR. Quá trình chỉnh sửa này đã tạo ra sự thay đổi từ Tryptophan (TGG) thành Leucine (TTG) tại amino acid 548 (W548L) và thay đổi từ Serine (AGT) thành Isoleucine (ATT) tại amino acid 627 (S627I), với tỷ lệ sửa chữa cao (16,88% HDR). Nghiên cứu đã chỉ ra rằng, chỉ cần một đột biến đơn W548L cũng đủ để dẫn đến tính kháng thuốc diệt cỏ BS [7]. Việc tạo ra dòng lúa kháng thuốc diệt cỏ nhanh chóng và hiệu quả ở nghiên cứu này còn cho thấy tiềm năng ứng dụng của hệ thống cgRNA/Cas9 trong chỉnh sửa gen ở các loài cây trồng khác. Ngoài ra, phương pháp Target-AID sử dụng phức hợp CRISPR/Cas9-cytidine deaminase cũng đã được áp dụng thành công để tạo ra tính kháng thuốc diệt cỏ Imazamox (IMZ) ở lúa (trong điều kiện nuôi cấy mô) thông qua tạo đột biến A96V trên ALS [8].
Thành công khác là gen ARM1(ARSENITE-RESPONSIVE MYB 1) – một gen thiết yếu trong việc điều chỉnh sự hấp thu và chuyển dịch Asen (As) từ rễ lên chồi trên cây lúa đã được chỉnh sửa để tạo đột biến bất hoạt gen (knock out) OsARM1 (OsARM1-KO) thông qua CRISPR/Cas9, giúp cải thiện đáng kể khả năng chống chịu As(III) ở lúa O. sativa Nipponbare (NPB), Dongjing (DJ) và SSBM [9]. Asen xuất hiện trong nhiều loại khoáng vật, được tìm thấy ở hai dạng vô cơ: arsenite [As(III)] và arsenate [As(V)]. Cả hai dạng Asen đều gây bất lợi đối với tế bào thực vật, As(III) liên kết với các nhóm sulfhydryl trong protein và ngăn chặn hoạt động của chúng, trong khi As(V) hoạt động như một chất tương tự phosphate làm ảnh hưởng đến một số quá trình sinh học thiết yếu, bao gồm tổng hợp ATP và phosphoryl hóa. Asen cũng là một chất gây ung thư nhóm I và là chất độc mạn tính, độc tính cao đối với con người, gây tổn thương da, gây bệnh tiểu đường… Tích lũy Asen trong gạo làm tăng sự tiếp xúc của con người với yếu tố gây ung thư độc hại này. Do vậy, giảm tích lũy Asen ở cây trồng nói chung và trên cây lúa nói riêng là điều cần thiết để tăng năng suất cây trồng và bảo vệ con người khỏi bị ngộ độc.
Đối với các gen liên quan đến các đặc tính năng suất (gen quy định tính trạng mùi hương hạt [10], ra hoa sớm [15], kiểu hình hạt [11]), dạng đột biến thường gặp là gây bất hoạt đơn hoặc đa gen thông qua cơ chế NHEJ. Ví dụ, việc loại bỏ gen Waxy ở lúa Xiushui 134 và Wuyunjing 7 đã tạo ra các dòng lúa (không mang gen chuyển) có hàm lượng amylose thấp hơn bình thường. Thông thường, gạo thương mại được phân loại thành 5 nhóm dựa vào hàm lượng amylose: nếp (0-5%), rất thấp (gạo dẻo) (5-12%), thấp (hơi dẻo) (12-20%), trung bình (20-25%) và cao (25-33%). Hàm lượng hàm lượng amylose càng thấp tức hàm lượng amylopectin trong thành phần tinh bột hạt gạo càng cao thì gạo càng dẻo. Từ đó, người ta phân biệt gạo nếp và gạo tẻ. Gạo nếp (0-5% amylose) có độ dính cao sau khi nấu chín, ngược lại amylose cao (25-33%) làm cơm khô, ít mềm [18]. Dòng lúa thu được trong nghiên cứu trên của Zhang và cộng sự rất có tiềm năng đối với ngành công nghiệp thực phẩm. Trong một số trường hợp khác, đột biến thay thế (amino acid hoặc alen) cũng đã được tạo ra thông qua cơ chế HR bởi hệ thống cgRNA/Cas9. Cụ thể, trong nghiên cứu gần đây của các nhà khoa học Trung Quốc, alen NRT1.1B trong các giống lúa thương mại đã được thay thế chính xác bằng alen ưu việt với mục tiêu nâng cao hiệu quả sử dụng đạm của cây lúa. Không cần tạo thêm áp lực chọn lọc bổ sung, alen NRT1.1B của giống lúa Japonica đã được thay thế bằng một alen từ giống lúa Indica chỉ trong một thế hệ với hiệu suất chỉnh sửa đạt 6,72% [17]. Công trình này cho thấy tính khả thi của việc thay thế bất cứ gen nào bằng các alen ưu việt trong một thế hệ, giúp tăng khả năng cải thiện các đặc điểm nông học quan trọng.
Bên cạnh Cas9 nuclease, Cpf1 nuclease cũng đã được sử dụng. Enzume Cpf1 là họ enzyme CRISPR nucleases loại V, bao gồm cả hoạt tính endoribonuclease và endodeoxyribonuclease. Do Cpf1 nhận biết trình tự bảo thủ protospacer (PAM) là 5-TTTN-3’ nên nó có thể được sử dụng để nhắm các mục tiêu giàu AT trong hệ gen. Hơn thế nữa, Cpf1 nucleases đã được chứng minh là có tỷ lệ chỉnh sửa ngoài mục tiêu thấp hơn so với Cas9 nuclease [19]. Vì thế, việc GE cây trồng bằng Cpf1 nucleases được cho là có tiềm năng tạo nên những tác động tích cực.
Thay lời kết
Công nghệ GE đang phát triển với tốc độ chóng mặt, đặc biệt hệ thống CRISPR/Cas đang tiến bộ nhanh hơn cả. Kể từ khi xuất hiện, CRISPR/Cas đã nhanh chóng chiếm lĩnh vị thế hàng đầu trong lĩnh vực khoa học thực vật, được ứng dụng ở hầu hết đối tượng cây trồng chính hiện nay. Ngoài hướng nghiên cứu cơ bản, CRISPR/Cas đã được ứng dụng để cải thiện một số đặc điểm định hướng thương mại, bao gồm các đặc tính nông học chính nhằm nâng cao chất lượng nông sản, tăng cường khả năng chống chịu stress, hay khả năng kháng thuốc diệt cỏ. Cây trồng chỉnh sửa bằng CRISPR/Cas9 được cho là vượt qua nhiều rào cản về xếp loại cây trồng biến đổi gen (Genetically Modified Crop – GMC) ở Hoa Kỳ, các nước thuộc vùng Scandinavi và khu vực châu Âu.
Thực phẩm biến đổi gen hiện nay vẫn là chủ đề gây tranh cãi trên phạm vi toàn cầu. Châu Âu vốn đã rất thận trọng trong việc cấp phép cho sản xuất đại trà GMC cũng như lưu hành các sản phẩm có nguồn gốc từ GMC trên thị trường. Đồng thời, một số nước trong số đó cũng bắt đầu cập nhật giải thích pháp lý về cây trồng GE [20]. Tuy nhiên, một thực tế đang được các nhà khoa học thừa nhận, đó là GE đã trở thành một công cụ đắc lực phục vụ chọn tạo giống cây trồng trong nền nông nghiệp chính xác, góp phần đảm bảo an ninh lương thực bền vững cho các nước hiện nay.
Để cây trồng GE được chấp nhận và các nghiên cứu GE trên cây trồng tiếp tục phát triển hiệu quả, thì sự thống nhất quốc tế về các quy định và định nghĩa về thực vật GE là rất cần thiết, qua đó thu hẹp khoảng cách giữa những rủi ro và tiềm năng của thực vật GE.
TÀI LIỆU THAM KHẢO
[1] A. Scheben, D. Edwards (2018), “Bottlenecks for genome-edited crops on the road from lab to farm”, Genome Biol., 19(1), p.178.
[2] A.M. Korotkova, et al. (2019), “Current achievements in modifying crop genes using CRISPR/Cas system”, Vavilov J. Genet. iBreed., 631(527), pp.224-234.
[3] T. Li, et al. (2012), “High-efficiency TALEN-based gene editing produces disease-resistant rice”, Nat. Biotech., 30, p.390.
[4] Q. Shan, et al. (2013), “Targeted genome modification of crop plants using a CRISPR-Cas system”, Nat. Biotechnol., 31, p.686.
[5] R. Mishra, R.K. Joshi, K. Zhao (2018), “Genome editing in rice: Recent advances, challenges, and future implications”, Front. Plant Sci., 9(1361), pp.1-12.
[6] K. Lu, et al. (2018), “Blocking amino acid transporter OsAAP3 improves grain yield by promoting outgrowth buds and increasing tiller number in rice”, Plant Biotechnol. J., 16(10), pp.1710-1722.
[7] H. Butt, et al. (2017), “Efficient CRISPR/Cas9-mediated genome editing using a chimeric single-guide RNA molecule”, Front. Plant Sci., 8, p.1441.
[8] Z. Shimatani, et al. (2018), “Herbicide tolerance-assisted multiplex targeted nucleotide substitution in rice”, Data Brief, 20, pp.1325-1331.
[9] F.Z. Wang, et al. (2017), “OsARM1, an R2R3 MYB transcription factor, is involved in regulation of the response to arsenic stress in rice”, Front. Plant Sci., 8(1868), pp.1-16.
[10] H.P. Lu, et al. (2017), “CRISPR-S: An active interference element for a rapid and inexpensive selection of genome-edited, transgene-free rice plants”, Plant Biotechnol. J., 15(11), pp.1371-1373.
[11] L. Shen, et al. (2017), “Rapid generation of genetic diversity by multiplex CRISPR/Cas9 genome editing in rice”, Sci. China Life Sci., 60(5), pp.506-515.
[12] Y. Sun, et al. (2017), “Generation of high-amylose rice through CRISPR/Cas9-mediated targeted mutagenesis of starch branching enzymes”, Front. Plant Sci., 8, pp.1-15.
[13] K. Abe, et al. (2018), “Production of high oleic/low linoleic rice by genome editing”, Plant Physiol. Biochem., 131, pp.58-62.
[14] M. Nieves-Cordones, et al. (2017), “Production of low-Cs(+) rice plants by inactivation of the K(+) transporter OsHAK1 with the CRISPR-Cas system”, Plant J., 92(1), pp.43-56.
[15] X. Li, et al. (2017), “High-efficiency breeding of early-maturing rice cultivars via CRISPR/Cas9-mediated genome editing”, J. Genet. Genomics, 44(3), pp.175-178.
[16] L. Tang, et al. (2017), “Knockout of OsNramp5 using the CRISPR/Cas9 system produces low Cd-accumulating indica rice without compromising yield”, Sci. Rep., 7(1), p.14438.
[17] J. Li, et al. (2018), “Efficient allelic replacement in rice by gene editing: A case study of the NRT1.1B gene”, J. Integr. Plant Biol., 60(7), pp.536-540.
[18] J. Zhang, et al. (2018), “Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of the Waxy gene in elite rice varieties”, J. Integr. Plant Biol., 60(5), pp.369-375.
[19] M.B. Begemann, et al. (2017), “Precise insertion and guided editing of higher plant genomes using Cpf1 CRISPR nucleases”, Sci. Rep., 7(1), p.11606.
[20] J. Metje-Sprink, et al. (2019), “DNA-free genome editing: Past, present and future”, Front. Plant Sci., 9(1957), pp.1-9.